4 Leggere e scrivere su file
Piccoli insiemi di dati possono essere gestiti con gli strumenti visti sino ad ora. Molto spesso abbiamo una varietà di fonti e di dati da gestire. In questo caso la maniera più semplice è quella di importare ed esportare i dati in file di testo. Il salvataggio dei dati in formato testo è particolarmente adatto ad essere utilizzato con altri programmi. Inoltre, si possono visionare i dati con un qualsiasi editor di testo anche se il programma che li ha originati non è più disponibile.
4.1 Posizione di lettura e scrittura
Uno degli aspetti più ostici che trovano i neofiti nell’uso di R è quello della gestione della posizione di lettura e scrittura di file nel filesystem del sistema operativo utilizzato, cioè in quale cartella (folder, directory) i file vengono letti o scritti. Per capire questi aspetti è importante conoscere:
- come si scrivono i percorsi nel proprio sistema operativo
- quali sono le funzioni R per interagire su proprio
Iniziamo illustrando due funzioni che servono per verificare la posizione predefinita (default) di scrittura e/o lettura dei file. La funzione per vedere quale sia la posizione predefinita è getwd (get working directory)
## [1] "/home/claudio/didattica/lezioni/psnotes/R/sito"da cui si vede che nel nostro caso la cartella di lavoro è /home/claudio/didattica/lezioni/psnotes/R/sito, si noti la sintassi tipica di un percorso nel sistema operativo Linux. Per cambiare la cartella predefinita di lettura/scrittura si usa la funzione setwd, ad esempio
che sposta la posizione nella cartella “/home/claudio/”.
4.1.1 Specificare percorsi
Vediamo ora come si scrivono i percorsi in tre diversi sistemi operativi. Vi consiglio di leggere anche come sono fatte le strutture delle cartelle. Vi ricordiamo che in tutti i sistemi operativi la struttura delle cartelle è ad albero, ad esempio una parte del mio albero è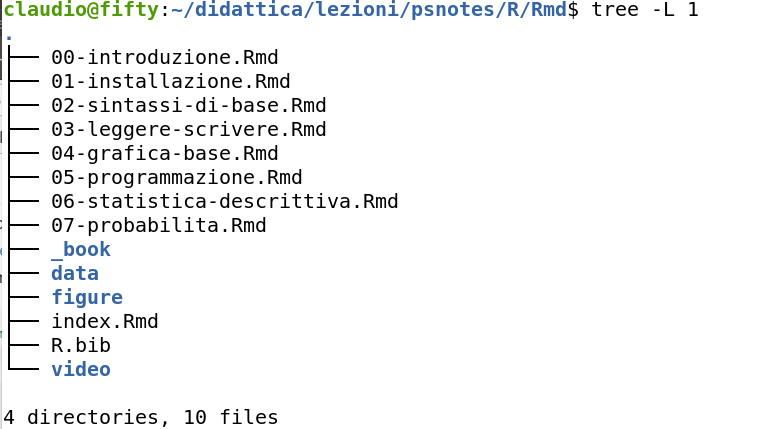
che ho ottenuto da R con il comando
dove quelli in nero sono file (ad esempio 01-installazione.Rmd), mentre in blu sono evidenziate le cartelle (ad esempio data).
4.1.1.1 Windows
Il percorso assoluto di solito inizia specificando l’unità fisica (o logica) su cui si vuole operare. L’unità predefinita è in genere indicata con C:\ e la separazione tra due nomi di cartelle è indicata con il simbolo \, ad esempio C:\cartella\sottocartella\. Quando però il percorso deve essere inserito nelle funzioni di R, per motivi che qui tralascio, il separatore deve essere raddoppiato, e quindi la sintassi è C:\\cartella\\sottocartella\\. Per evitare il raddoppio, solo all’interno delle funzioni R, si può utilizzare come separatore il simbolo /, cioè possiamo scrivere C:/cartella/sottocartella/. Nel caso di percorsi relativi (cioè che si riferiscono alla posizione attuale), il percorso deve iniziare con .\\ (oppure ./), che significa dalla posizione corrente, oppure ..\\ (oppure ../), che significa dalla posizione di livello superiore nell’albero. Ovviamente questi simboli possono essere ripetuti, ad esempio ..\\..\\ (oppure ../../) significa posizionarsi due posizioni sopra nella struttura ad albero.
4.1.1.2 Unix-like
Per sistemi operativi Unix-like, ad esempio GNU/Linux o Mac OS la sintassi per i percorsi è la stessa, il percorso assoluto inizia con la radice dell’albero / seguita dal nome delle cartelle separate dal simbolo /, ad esempio /home/claudio/ indica come posizione la mia cartella personale come utente nel sistema. Nel caso di percorsi relativi possiamo usare la sintassi ./, ../ etc. in modo analogo al caso dei sistemi Windows.
4.1.1.3 Specificare percorsi con R (progredito)
Vi sono alcune funzioni che possiamo usare per gestire i percorsi in maniera più semplice, automatica e indipendente dal sistema operativo. Queste sono file.path, path.expand, basename e dirname. Supponiamo, ad esempio di voler creare il percorso per un file, che nella sintassi unix-like è /home/claudio/didattica/prova.R possiamo scrivere, salvando il risultato nell’oggetto percorsocompleto,
## [1] "/home/claudio/didattica/prova.R"con la funzione basename otteniamo il solo nome del file
## [1] "prova.R"mentre con dirname otteniamo il solo percorso
## [1] "/home/claudio/didattica"4.1.1.4 Esercizio
- Scoprire come si chiama la vostra cartella personale nel vostro sistema operativo. Questa può differire dal nome visto nelle interfaccie grafiche, ad esempio, nell’interfaccia il nome di una cartella è in italiano mentre all’interno del filesystem è in lingua inglese. Per Windows potete usare la shell o la powershell. Per sistemi Unix-like potete usare il terminale
- Scoprire la vostra cartella di lavoro in R, nel momento in cui accedete ad R. In questo caso è sufficiente usare la funzione
getwd() - Cambiare la cartella di lavoro posizionandola nella vostra cartella personale usando la funzione
setwde controllare con la funzionegetwd()che l’operazione sia avvenuta correttamente.
4.1.2 Convenzione per il nome dei file e delle cartelle
E’ opportuno leggere la convenzione dei nomi per capire quali nome dei file possiamo usare, a seconda del sistema operativo che si utilizza. Tuttavia è buona prassi usare nome dei file (e delle cartelle) che possano essere validi in tutti i sistemi operativi e in particolare:
- non si usano spazi all’interno dei nomi dei file
- non si usano caratteri speciali (accenti,
\slash e/backslash, etc.) all’interno dei nomi dei file
- si suggerisce di evitare anche l’uso del carattere
_(sottolineato) e-(trattino)
Le stesse regole dovrebbero essere utilizzate per i nomi delle cartelle.
4.1.3 Gestione dei file da R (progredito)
In alcuni casi è necessario verificare direttamente da R la presenza o meno di un file in una certa cartella, oppure rinominarlo o cancellarlo. Per compiere queste operazioni possiamo usare delle funzioni che come parte iniziale del loro nome hanno file.. Ad esempio
## [1] FALSE## [1] TRUE## [1] TRUEper prima cosa abbiamo controllato che il file non esistesse, poi lo abbiamo creato e infine abbiamo controllato nuovamente la sua esistenza. Ovviamente il file creato è vuoto e risiede nella cartella di lavoro predefinita. Adesso rimuoviamo il file
## [1] TRUE## [1] FALSEPossiamo vedere tutti i file in una certa cartella (o le sue sottocartelle) con le funzioni
## [1] "_book" "_main.Rmd"
## [3] "00-introduzione.Rmd" "01-installazione.Rmd"
## [5] "02-sintassi-di-base.Rmd" "03-leggere-scrivere.Rmd"
## [7] "04-grafica-base.Rmd" "05-programmazione.Rmd"
## [9] "data" "esempio.txt"
## [11] "figure" "index.Rmd"
## [13] "R.bib" "video"## [1] "."
## [2] "./_book"
## [3] "./_book/figure"
## [4] "./_book/libs"
## [5] "./_book/libs/gitbook-2.6.7"
## [6] "./_book/libs/gitbook-2.6.7/css"
## [7] "./_book/libs/gitbook-2.6.7/css/fontawesome"
## [8] "./_book/libs/gitbook-2.6.7/js"
## [9] "./_book/libs/jquery-2.2.3"
## [10] "./_book/video"
## [11] "./data"
## [12] "./figure"
## [13] "./video"ed infine possiamo creare o verificare l’esistenza di una cartella con le funzioni dir.create e dir.exists.
4.1.4 Formati dei file
La distinzione principale che possiamo fare è tra file in formato testo (chiamati spesso anche in formato ASCII) e quelli in formato binario. Mentre i file in formato testo sono “facilmente” leggibili da programmi diversi, quelli in formato binario hanno una struttura che è spesso specifica per ogni programma/sistema che si utilizza. Per questo molto spesso i file di testo sono utilizzati per archiviare dati “importanti” che si vuole poter leggere con sicurezza anche in futuro, viceversa i file binari sono utilizzati per il lavoro corrente. In R ci sono funzioni apposite per poter scrivere file di diversi formati, sia in formato testo che binario. Nella sezione successiva ne illustreremo le principali.
4.2 Esempio introduttivo sulla lettura di file
Quasi sempre l’insieme di dati con cui dobbiamo lavorare è abbastanza grande da rendere la digitazione dei dati ogni volta che ne abbiamo bisogno una operazione lunga e non priva di errori. Per questo motivo, i dati possono essere salvati all’interno di un file e poi associati ad un oggetto di R attraverso alcune funzioni.
I dati possono essere scritti ad esempio con un editor di testo e salvati in modalità testo (ASCII): ogni singolo dato deve essere separato dal successivo con un qualche separatore, ad esempio uno spazio (Altri separatori utilizzati spesso sono la virgola e il punto e virgola). Si supponga che il file di dati si chiami provincie.csv con il contenuto
Pop.f, Pop.m, Iscritti
420319, 394262, 19789,
497319, 456654, 32371,
110007, 101041, 4871e sia disponibile in un qualche posto nel web, ad esempio sul sito del corso con indirizzo
allora lo possiamo leggere attraverso la funzione read.table
## Pop.f Pop.m Iscritti
## 1 420319 394262 19789
## 2 497319 456654 32371
## 3 110007 101041 4871specificando che la prima riga contiene l’intestazione (header=TRUE) e che il separatore utilizzato è la virgola (sep=","). Con la funzione count.fields otteniamo per ogni colonna il numero di osservazioni disponibili
## [1] 3 3 3 3Supponiamo ora di aggiungere al data.frame la variabile Cancellati e di voler salvare in un file il nuovo insieme di dati usando la funzione write.table
Cancellati <- c(18187, 27460, 4871)
write.table(x=cbind(provincie.dati, Cancellati),
file="./data/provincie.nuovo.csv", sep=",", quote=FALSE, row.names=FALSE)E’ possibile salvare in un file l’intera sessione di lavoro o parte di essa con la funzione save.image e save, mentre con la funzione load si può caricare gli oggetti o la sessione di lavoro salvata
## [1] "Cancellati" "cn" "CO2" "l"
## [5] "M" "M1" "percorsocompleto" "posizione"
## [9] "string" "v" "value" "value1"
## [13] "value2" "valueInt" "w" "x1"
## [17] "X1" "x2" "X2" "x3"
## [21] "y" "y2"## [1] "Cancellati" "cn" "CO2" "l"
## [5] "M" "M1" "percorsocompleto" "posizione"
## [9] "provincie.dati" "string" "v" "value"
## [13] "value1" "value2" "valueInt" "w"
## [17] "x1" "X1" "x2" "X2"
## [21] "x3" "y" "y2"4.3 Funzioni per la scrittura e la lettura su file
Ci sono diverse funzione che servono per esportare (scrivere) e importare (leggere) dati da/in R. Le elenchiamo qui di seguito e successivamente descriviamo l’uso di alcune di esse.
catscrive gli oggetti di R a terminale (li stampa, analogamente alla funzioneprint) oppure li scrive in un file, è la funzione di basso livello per la scrittura dei dati su file
scanlegge i dati e li trasfroma in vettori o liste, è la funzione di basso livello per la lettura dei dati da fileread.table,read.csv, per leggere dati che sono salvati in forma di tabella e/o utlizzando come separatore tra i campi la virgola (la funzione inversa è \write.table`read.fwfpuò essere utilizzato per leggere dati salvati in formato fissoreadLines, per leggere righe di un file di testo (la funzione inversa èwriteLines)source, leggere codice R ed eseguirlo (la funzione inversa èdump)dget, per leggere file che contengono oggetti R (la funzione inversa èdput)load, per leggere un spazio di lavoro (workspace) (le funzioni inverse sonosaveesave.image)unserialize, per leggere singoli oggetti R in forma binaria
Per leggere da file in formato spreadsheet (ad esempio da openoffice o office), la maniera più semplice è quella di salvare il foglio di lavoro (worksheet) in formato csv (comma separated file) e poi leggerlo con la funzione read.csv. Un secondo modo è quello di aprire una connessione direttamente nel formato originale attraverso il protocollo ODBC. Questo risulta in genere più complicato.
4.3.1 write.table e read.table
Cominciamo a descrivere la funzione write.table che viene utilizzata principalmente per scrive oggetti di classe data.frame, matrix e vector in un file di nome specificato. Uno spazio è utilizzato per separare i campi quando sep=" " è specificato tra gli argomenti della funzione. Altre possibilità includono la virgola (sep=","), o il tabulatore (sep="\t"). Usiamo ad esempio l’insieme di dati CO2 direttamente disponibile in R usando la funzione data
e creiamo una cartella di nome data a partire dalla cartella di lavoro in cui siamo (A proposito in quale cartella di lavoro siamo? Potresti usare la funzione getwd() per vedere …). Usando la funzione str possiamo avere un’idea di come è fatto questo oggetto, che contiene un insieme di dati
## Classes 'nfnGroupedData', 'nfGroupedData', 'groupedData' and 'data.frame': 84 obs. of 5 variables:
## $ Plant : Ord.factor w/ 12 levels "Qn1"<"Qn2"<"Qn3"<..: 1 1 1 1 1 1 1 2 2 2 ...
## $ Type : Factor w/ 2 levels "Quebec","Mississippi": 1 1 1 1 1 1 1 1 1 1 ...
## $ Treatment: Factor w/ 2 levels "nonchilled","chilled": 1 1 1 1 1 1 1 1 1 1 ...
## $ conc : num 95 175 250 350 500 675 1000 95 175 250 ...
## $ uptake : num 16 30.4 34.8 37.2 35.3 39.2 39.7 13.6 27.3 37.1 ...
## - attr(*, "formula")=Class 'formula' language uptake ~ conc | Plant
## .. ..- attr(*, ".Environment")=<environment: R_EmptyEnv>
## - attr(*, "outer")=Class 'formula' language ~Treatment * Type
## .. ..- attr(*, ".Environment")=<environment: R_EmptyEnv>
## - attr(*, "labels")=List of 2
## ..$ x: chr "Ambient carbon dioxide concentration"
## ..$ y: chr "CO2 uptake rate"
## - attr(*, "units")=List of 2
## ..$ x: chr "(uL/L)"
## ..$ y: chr "(umol/m^2 s)"Come potete vedere è un oggetto piuttosto complicato perché ha diverse classi (classes) ma tra queste vi è anche data.frame. E’ formato da 84 osservazioni (righe) e 5 variabili (colonne). Usiamo la funzione data.matrix per ottenere un oggetto di tipo matrix (questa operazione non è necessaria, ma rende più semplice capire quello che stiamo facendo)
## num [1:84, 1:5] 1 1 1 1 1 1 1 2 2 2 ...
## - attr(*, "dimnames")=List of 2
## ..$ : chr [1:84] "1" "2" "3" "4" ...
## ..$ : chr [1:5] "Plant" "Type" "Treatment" "conc" ...## [1] TRUE## [1] FALSEUsiamo la funzione write.table per scrivere un file in formato testo, nella cartella ./data, che contenga l’oggetto co2, in cui le variabili siano separate da ; e le osservazioni siano su righe diverse
Dall’altra parte, read.table legge da un file di testo esterno e crea un oggetto data.frame. Ad esempio, dato che la prima riga di testo (il primo record) del file CO2.txt consiste nel nome delle variabili, il seguente comando ci darà il risultato desiderato
dove con header=TRUE abbiamo indicato che la prima riga contiene il nome delle variabili. Notate che l’oggetto restituito non è un oggetto di tipo matrix ma di tipo data.frame
## [1] FALSE## [1] TRUEOvviamente avremmo potuto gestire il percorso anche in maniera diversa. Ad esempio
## [1] "/home/claudio/didattica/lezioni/psnotes/R/sito"in questa maniera la nuova cartella di lavoro è
## [1] "/home/claudio/didattica/lezioni/psnotes/R/sito//data/"e quindi possiamo leggere direttamente il file
ora riportamo la cartella di lavoro nella sua posizione precedente
Ricapitolando, per insiemi di dati di dimensioni piccole, generalmente si può richiamare la funzione read.table utilizzando i valori predefiniti per gli argomenti della funzione. R ignorerà automaticamente tutte le righe che cominciano con #, conterà quante righe sono presenti (e quanta memoria sarà necessario riservare), individuerà quale tipo di variabili ci sono in ogni colonna della tabella. Se però forniamo a R tutte queste informazioni la funzione verrà eseguita più velocemente e in maniera più efficiente. read.csv coincide con read.table tranne che il valore predefinito del separatore è la virgola, mentre read.csv2 è utile quando viene usata la virgola come segno decimale per i numeri e si usa come separatore il punto e virgola. In maniera analoga vi è la funzione read.delim che utilizza come separatore il tabulatore (TAB, \t).
Per insiemi di dati di dimensione più grande, le seguenti cose renderanno la vostra vita più facile. Leggi la pagina di aiuto della funzione read.table, che contiene molti consigli utili. Fai un calcolo approssimato della quantità di memoria necessaria per gestire il tuo insieme di dati. Se il tuo insieme necessita di una memoria superiore a quella disponibile nel tuo computer è necessario utilizzare procedure speciali, alcuni consigli sono disponibili ad esempio qui. Poni l’argomento comment.char = "" se non ci sono linee di commento presenti nel tuo file.
Per scrivere insiemi di dati di dimensioni grandi, write.matrix (nel pacchetto MASS) è in genere più efficiente in termini di gestione della memoria rispetto alla funzione write.table. I dati possono essere scritti in blocchi usando l’argomento blocksize come mostrato qui di seguito
## Loading required package: MASS4.3.2 cat e scan
Le funzioni cat e scan sono particolarmente utili per la scrittura e la lettura di oggetti con una certa complessità in formato testo. Entrambe possono interagire con la console, cioè, la prima può stampare sulla console, la seconda può ottenere dati dalla console. Ad esempio
notate che quando eseguirete questo pezzo di codice, diversamente da quello che vedete qui, il “prompt” tra i due comandi non è andato a capo e le due stringhe verranno stampate una dietro l’altra, seguite dal simbolo >. Possiamo scrivere
## Prova1
## Prova2così facendo avremo invece che saremo su di una riga nuova ogni volta, infatti \n vuol dire new line. Per scrivere il contenuto in un file di testo, basta specificare il nome del file
possiamo leggere il file con la funzione scan
## [1] 2 3 5 7 11 13 17dove con l’argomento skip abbiamo saltato la lettura della prima riga (del primo record). Si possono anche specificare il numero di righe da leggere
## [1] 2 3 5 74.4 Interfacce con il mondo esterno (progredito)
I dati possono essere letti attraverso connessioni (connection interfaces). Le connessioni, nella maggior parte dei casi, sono con file ma in alcuni casi con oggetti più complicati.
file, apre una connessione con un filegzfile, apre una connessione con un file compresso in formatogzipbzfile, apre una connessione con un file compresso in formatobzip2url, apre una connessione con un sito web.
4.4.1 Connessioni
Cominciamo con vedere come aprire connessioni a file
## function (description = "", open = "", blocking = TRUE, encoding = getOption("encoding"),
## raw = FALSE, method = getOption("url.method", "default"))description è il nome del file, open indica il modo con cui viene aperta una connessione: “r” sola lettura (read only), “w” solo scrittura (writing) e inizializzazione di un nuovo file, “a” aggiungi (appending) “rb”, “wb”, “ab” leggere, scrivere o aggiungere in formato binario (Windows).
In generale le connessioni sono strumenti potenti per utilizzare file e altri oggetti esterni. In pratica, nella maggior parte dei casi, non abbiamo bisogno di lavorare direttamente con connessioni. Cioè
è lo stesso di
4.4.2 Importare file binari
Dati in formato binario sono spesso scritti da programmi di analisi statistica dei dati e possono essere letti da R, anche se si suggerisce di utilizzare i formati di R quando ciò sia possibile. Il pacchetto R foreign rende disponibili diverse funzioni per importare dati da: EpiInfo, Minitab, S-Plus, SAS, SPSS, Stat e Systat. Qui, una lista delle funzioni
read.epiinfolegge i file in formato EpiInforead.mtpimporta i fogli di lavoro (worksheets) di Minitabread.xportlegge i file in formato SAS nel formato TRANSPORTread.Slegge i file binari prodotte da S-PLUS 3.x, 4.x or 2000 su (32-bit) Unix o Windows. data dumps da S-PLUS 5.x r 6.x usando la funzione (di S-PLUS)dump(..., oldStyle=T)possono essere letti con la funzionedata.restoreread.spsslegge i file in formato SPSS creati con le funzionisaveeexportread.dtalegge i file in formato Stataread.systatleggei file in formato Systat
4.4.3 Salvare i dati in formato “non-tabella”
Per un’archiviazione temporanea o per il trasporto (da un computer ad un altro) è più efficiente salvare i dati in formato binario (compresso) usando le funzioni save o save.image.
x <- 1
y <- data.frame(a=1, b="a")
save(x, y, file = "./data/esempio.RData")
load("./data/esempio.RData") ## sovrascrive gli oggetti x e y!L’uso di formati binari non è molto adatto per conservare i file per lungo tempo perché nel caso vengano corrotti non è possibile recuperarli.
4.4.4 Deparsing oggetti R
Un’altro modo per conservare dati è quello di utilizzare il deparsing degli oggetti R con la funzione dput e la loro lettura usando la funzione dget.
## structure(list(a = 1, b = "a"), class = "data.frame", row.names = c(NA,
## -1L))dove il risultato mostra l’oggetto una volta che viene scritto direttamente in codice R (l’operazione viene chiamata deparsing). Notate l’uso della funzione structure, ad esempio, possiamo creare un oggetto R nel modo seguente
structure(list(a = 1,
b = structure(1L, .Label = "a",
class = "factor")),
.Names = c("a", "b"), row.names = c(NA, -1L),
class = "data.frame")## a b
## 1 1 ain modo che oltre al contenuto abbiano anche degli attributi, ma questo è un argomento che non affrotiamo. Riassumendo possiamo scrivere in un file in formato testo l’oggetto y
e successivamente leggerlo
## a b
## 1 1 a4.4.5 Dumping oggetti di R
Se vogliamo salvare molti oggetti, in formato testo, possiamo usare la funzione dump
x <- "foo"
y <- data.frame(a = 1, b = "a")
dump(c("x", "y"), file = "./data/esempio.Rdump")
rm(x, y)e poi leggerli utilizzando la funzione source
## a b
## 1 1 a## [1] "foo"Le funzioni dump e dput che scrivono in un file oggetti in formato testo, consetono di modificare gli oggetti direttamente nel file attraverso un editor di testo e in caso di corruzione del file risulta più semplice recuperare il contenuto. Diversamente dalla funzioni che scrivono i dati in formato di “tabella” o in un file csv, queste due funzioni conservano tutti i metadati (gli attributi). Infine i file in formato testo funzionano molto bene quando si usano programmi che tengono traccia dei cambiamenti, come ad esempio git, infine i file di testo sono conformi alla Unix philosophy.
4.4.6 Leggere righe di un file di testo
La funzione readLines può essere usata per leggere righe di un file di testo. Ad esempio, per prima cosa creiamo un file compresso in formato testo usando una connessione
successivamente, apriamo nuovamente la connessione e utilizziamo la funzione readLines per leggere le prime 10 righe
## [1] "1 1 1 1 1" "1 1 2 2 2" "2 2 2 2 3" "3 3 3 3 3" "3 4 4 4 4" "4 4 4 6 6"
## [7] "6 6 6 6 6" "5 5 5 5 5" "5 5 9 9 9" "9 9 9 9 8"in maniera analoga possiamo aprire una connessione di testo con la funzione textConnection
e leggere il suo contenuto con la funzione read.table
## X1.1.1.1.1
## 1 1 1 2 2 2
## 2 2 2 2 2 3
## 3 3 3 3 3 3
## 4 3 4 4 4 4
## 5 4 4 4 6 6
## 6 6 6 6 6 6
## 7 5 5 5 5 5
## 8 5 5 9 9 9
## 9 9 9 9 9 8Infine la funzione writeLines può essere usata per scrivere in un file un vettore composto da caratteri.
La funzione readLines può essere usata anche per leggere il contenuto di pagine web, che dovrà essere successivamente processato per poter essere analizzato.
## Warning in readLines(con): incomplete final line found on 'http://www.unitn.it'## [1] ""
## [2] "<!DOCTYPE html>"
## [3] "<html lang=\"it\" dir=\"ltr\">"
## [4] "<head>"
## [5] " <!--[if IE]><![endif]-->"
## [6] "<meta http-equiv=\"Content-Type\" content=\"text/html; charset=utf-8\" />"